Tecnología del año: Transcriptómica resuelta espacialmente.
La transcriptómica resuelta espacialmente ha sido coronada como la tecnología del año por Nature Methods, gracias a este método revolucionario los investigadores tienen acceso a la transcriptómica además de conocer el contexto espacial de las células del tejido en estudio.
La solución VISIUM Spatial Gene Expression de 10x Genomics cuantifica el ARNm total en secciones de tejido intacto y mapea las ubicaciones donde se produce la actividad genética. Esta tecnología se basa en el diseño de slides con áreas de captura que a su vez presentan puntos de expresión génica denominados spots. Estos spots contienen los cebadores necesarios para la captura de los ARNm poli-adenilados.
Las secciones de tejido se colocan en estas áreas de captura; posteriormente son fijadas y teñidas con Hematoxilina y Eosina o mediante inmunofluorescencia, como se describe en los protocolos demostrados de fijación y tinción de tejidos – Methanol Fixation, H&E Staining & Imaging for Visium Spatial Protocols y Methanol Fixation, Immunofluorescence Staining & Imaging for Visium Spatial Protocols, respectivamnte . Una vez teñidos y tomada la imagen de cada una de las áreas de captura, se lleva a cabo la permeabilización del tejido liberando su ARNm, el cual es capturado por las secuencias poli(dT) ubicados en los spots. Todo el cDNA generado a partir del ARNm capturado por cebadores en un lugar específico comparte una secuencia 10x Barcode la cual permite ubicar la procedencia del transcrito dentro de la morfología del tejido.
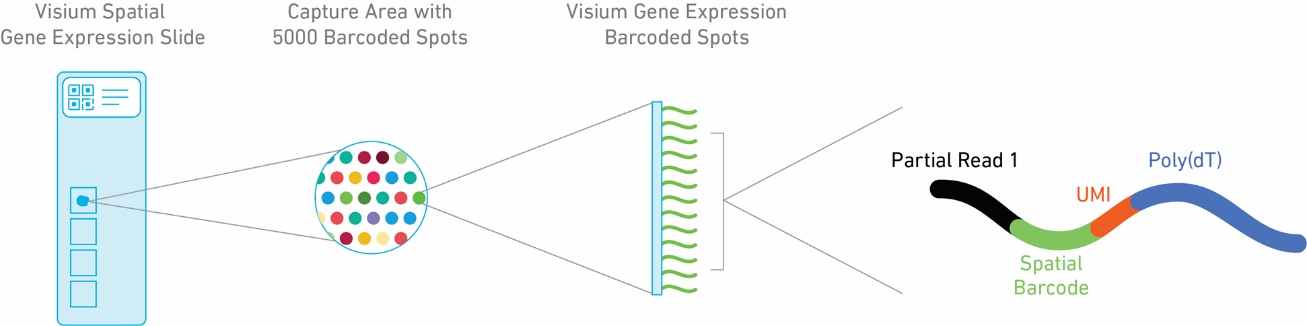
El slide incluye 4 áreas de captura (6,5 x 6,5 mm), cada una definida por un marco denominado fiducial frame (fiducial frame + área de captura es de 8 x 8 mm). Estas áreas de captura presentan ~ 5000 spots, cada cualcon cebadores que incluyen (Figura 1):
- Illumina TruSeq Read 1 (cebador de secuenciación de lectura parcial 1)
- Código de barras espacial de 16 nt (todos los cebadores de un mismo spot comparten la misma secuencia 10x Barcode)
- 12 nt identificador molecular único (UMI)
- Secuencia poli (dT) de 30 nt (captura el ARNm poliadenilado para la síntesis de cDNA
Figura 1. Spatial Gene Expression Slide.
Existen diferentes tejidos ya validados para la tecnología VISIUM Spatial Transcriptomics listados en el siguiente enlace: Visium Spatial Gene Expression Optimized Tissues . A día de hoy, se trabaja a partir de muestras de tejido fresco-congelado y embebido en OCT. Las condiciones de preparación, crioseccionado y almacenaje quedan recogidas en el siguiente documento Visium Spatial Protocols – Tissue Preparation Guide. Una vez colocados las secciones de tejido en las respectivas áreas de captura, estas pueden ser almacenados a -80oC hasta su procesamiento. En cuanto a las dimensiones de los cortes de tejidos, vienen delimitados por el tamaño del área de captura (6.5mmx6.5mm), mientras que el grosor recomendado se encuentra entre los 10 y 20um dependiendo de la naturaleza la muestra.
Previo a comenzar con el ensayo Gene Expression, es necesario determinar el tiempo óptimo de permeabilización para cada tipo de muestra en concreto. Paso necesario en caso de que se cambien las condiciones de preparación del tejido o las condiciones de tratamiento del experimento. Para ello, 10x Genomics ha diseñado un segundo slide con 8 áreas de captura donde permite testar hasta 6 tiempos de permeabilización distintos para un mismo tipo de tejido. En este caso, el cDNA sintetizado a partir de los ARNm liberados deja una huella fluorescente que será captura por microscopia. Está imagen será evaluada en base a la intensidad y difusión con el objetivo de establecer el tiempo de permeabilización óptimo. El proceso queda descrito en el siguiente documento Visium Spatial Tissue Optimization Reagents Kits User Guide.
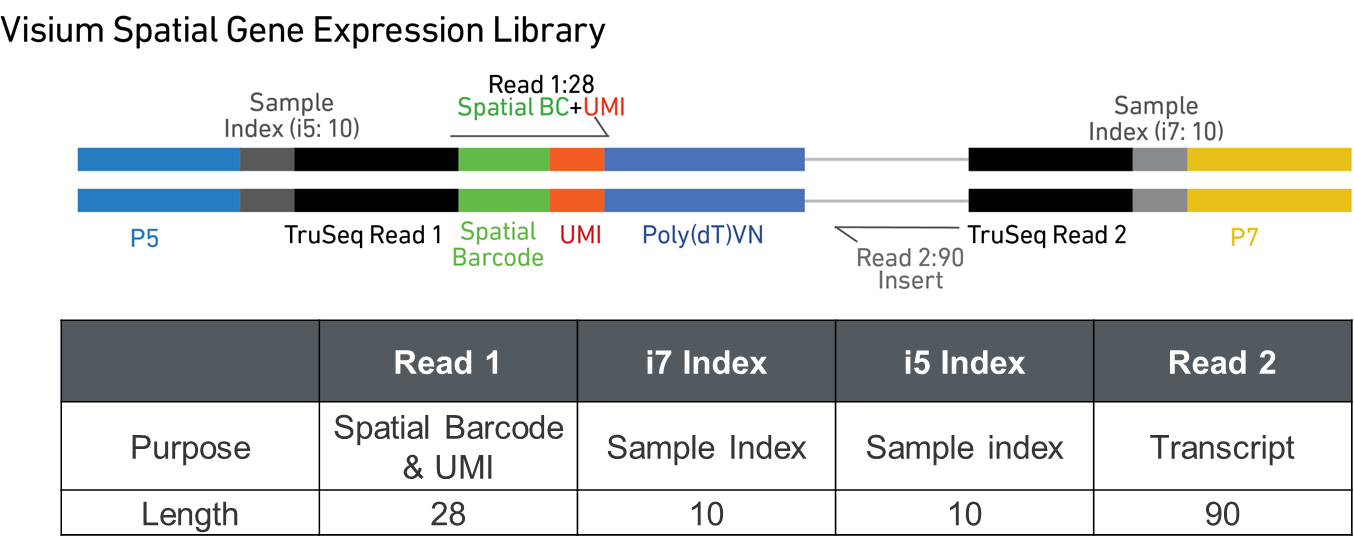
Las bibliotecas generadas por la solución Visium Spatial Gene Expression, cuyo protocolo está descrito en el siguiente documento Visium Spatial Gene Expression Reagent Kits User Guide, comprende construcciones pair-end compatibles con los secuenciadores Illumina, flanqueadospor los adaptadores P5 y P7. La profundidad de lectura recomendada es de 50.000 pares de lectura por spot cubierto por tejido, en cuanto a las especificaciones vienen descritas en la figura 5:
Figura 5. Librerías Visium Saptial Gene Expression
Los archivos de datos BCL generados serán analizados por el software Space Ranger, disponible en la página web de 10xGenomics. Space Ranger es un conjunto de pipelines de análisis que procesan los output del RNA-Seq para alinear las lecturas, generar matrices y realizar análisis de agrupación y expresión génica. Space Ranger incluye 2 pipelines relevantes para los experimentos de expresión génica espacial:
- Cellranger mkfastq que demultiplexa los archivos BCL generados por secuenciadores de Illumina en archivos FASTQ; y
- Cellranger count, este toma los archivos FASTQ y realiza la alineación (STAR), filtrado y cuantificación de secuencias barcode y UMI. Utiliza las secuencias barcode y UMI para generar matrices sobre los que realiza los análisis secundarios: Análisis de Principales componentes para reducir dimensionalidad de los datos; los algoritmos Graph-Based and K-Means para la clusterización de los datos y sSeq para el estudio de expresión diferencial de genes.
Finalmente se podrán visualizar los datos con el software Loupe Browser de 10xGenomics.