La solución VISIUM Spatial Gene Expression de 10x Genomics es una tecnología que permite obtener el transcriptoma resuelto espacialmente y ha sido coronada como la tecnología del año por Nature Methods. Visium permite a los investigadores tener acceso a la transcriptómica además de conocer el contexto espacial de las células del tejido en estudio.
De un modo muy general, Visium se basa en el diseño de slides con áreas de captura donde se colocan las secciones de tejido. Cada área de captura tiene al redor de 5 mil puntos de expresión génica denominados spots. Una vez en esa área de captura, el corte de tejido es teñido, con N&E o con inmunofluorescencia. Una vez teñidos y tomada la imagen de cada una de las áreas de captura, se lleva a cabo la permeabilización del tejido liberando sus ARNm, los cuales son capturado por las secuencias poli(dT) ubicadas en los spots. Todo el cDNA generado a partir del ARNm capturado por cebadores en un lugar específico comparte una secuencia 10x Barcode la cual permite ubicar la procedencia del transcrito dentro de la morfología del tejido.
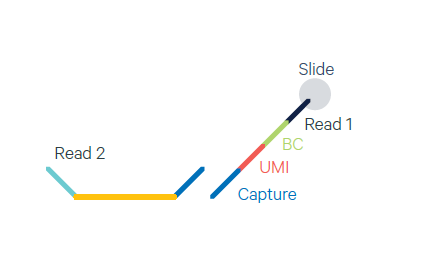
Como se puede comprobar en la Figura 1, el slide incluye 4 áreas de captura (6,5 x 6,5 mm), cada una definida por un marco denominado fiducial frame (fiducial frame + área de captura es de 8 x 8 mm). Estas áreas de captura presentan ~ 5000 spots, cada cual con cebadores que incluyen:
- Illumina TruSeq Read 1 (cebador de secuenciación de lectura parcial 1)
- Código de barras espacial de 16 nt (todos los cebadores de un mismo spot comparten la misma secuencia 10x Barcode)
- 12 nt identificador molecular único (UMI)
- Secuencia poli (dT) de 30 nt (captura el ARNm poliadenilado para la síntesis de cDNA)
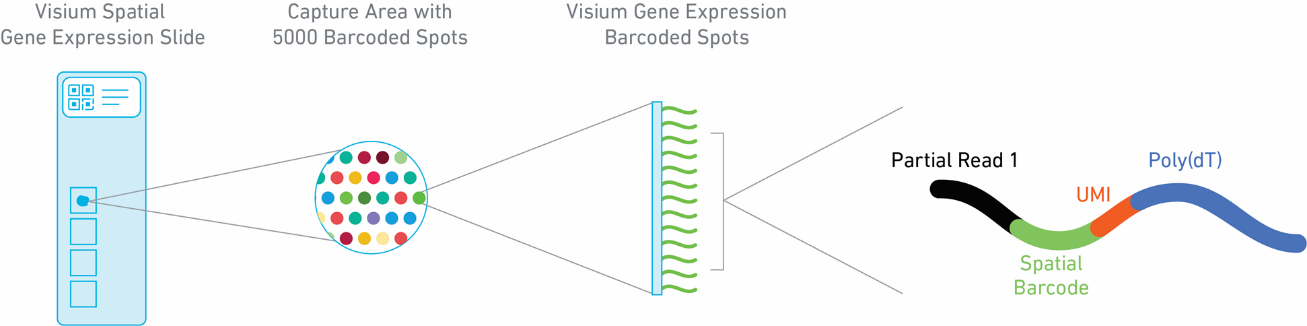
Figura 1. Spatial Gene Expression Slide.
Hasta ahora Visium solo era compatible con tejido fresco-congelado y embebido en OCT, pero 10X Genomics acaba de lanzar la solución Visium compatible con muestras procesadas en FFPE (tejidos fijados en formalina y embebidos en parafina).
La conservación de los tejidos en FFPE es el método más común para preservar el tejido, pero el procesamiento en FFPE puede dañar significativamente las moléculas de ARN, complicando el estudio del transcriptoma. El lanzamiento de la nueva solución Visium compatible con FFPE es una tecnología innovadora que complementa el análisis patológico tradicional al combinar los beneficios de las técnicas histológicas con la obtención del transcriptoma de una manera eficiente
Visium para FFPE utiliza los mismos slides y los mismos cebadores que Visium para FF (tejido fresco congelado) pero tiene una nueva tecnología para garantizar la captura de los ARNm que consiste en utilizar pares de sondas capaces de identificar y hibridar con los ARNm posibilitando así una detección altamente especifica y sensible de todo el transcriptoma en cortes de tejido FFPE de humano o de ratón.
En la figura 2 se encuentran esquematizados los pasos del flujo de trabajo para la construcción de las librerías.
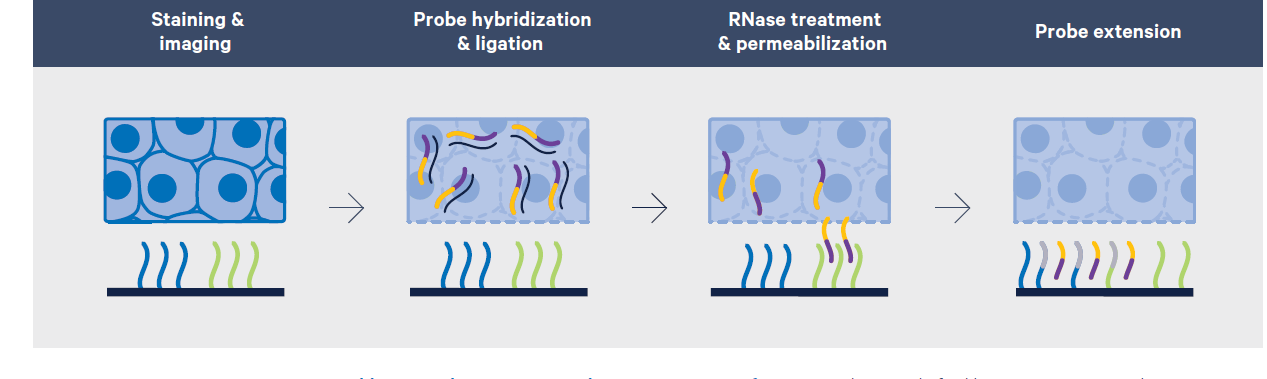
Figura 2. Construcción de las librerías para Visium FFPE
Tinción e imágenes
Los tejidos FFPE se seccionan y se colocan en las áreas de captura del slide. Utilizando técnicas de tinción estándar, que incluyen tinción con H&E o inmunofluorescencia, los cortes de tejido se visualizan en el slide con un microscopio.
Hibridación y ligación de las sondas
A continuación, el tejido se desreticula para liberar el ARNm que fue secuestrado por la fijación con formalina. Luego el tejido se incuba con un panel de sondas para el transcriptoma completo de humano o ratón, que consiste en un par de sondas específicas y pre-diseñadas para cada gene (~18 mil genes de humano/~20 mil genes de ratón). Estos pares de sondas se hibridan con su gen (Figura 3) y luego se ligan entre sí.
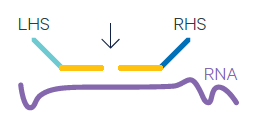
Figura 3. Par de sondas pre-diseñadas para hibridar con secuencias específicas de RNA
Permeabilización y tratamiento con la RNasa
Los pares de sonda ligadas entre si se liberan del tejido tras el tratamiento con RNasa y la permeabilización del tejido. Los pares de sondas ligados se unen con los cebadores (Figura 4) que se encuentran en el área de capturas y que contienen la información espacial.
Figura 4. Par de sondas ligadas y capturadas por los cebadores fijados en el área de captura del slide
Extensión de las sondas RTL (RNA-templated ligation)
Los pares de sondas se amplían para incorporar las distintas secuencias de nucleótidos que se encuentran en los cebadores. El producto final se libera del slide y se amplifica con una PCR. De seguida, los productos son procesados hasta que se obtenga una biblioteca de expresión genética espacial que se puede secuenciar utilizando secuenciadores de lectura corta. La profundidad de lectura recomendada es de 25.000 pares de lectura por spot cubierto por tejido.
Para finalizar, los datos se procesan y visualizan utilizando el software de análisis Space Ranger y el software de visualización Loupe Browser, ambos gratis para descargar en la página de 10X Genomics.
Para finalizar, los datos se procesan y visualizan utilizando el software de análisis Space Ranger y el software de visualización Loupe Browser, ambos gratis para descargar en la página de 10X Genomics.
Hoy en día existen diferentes tejidos tanto de ratón (Fig. 5) como de humano (Fig. 6) ya validados para esta tecnología, sin embargo, aún no está disponible una lista completa.
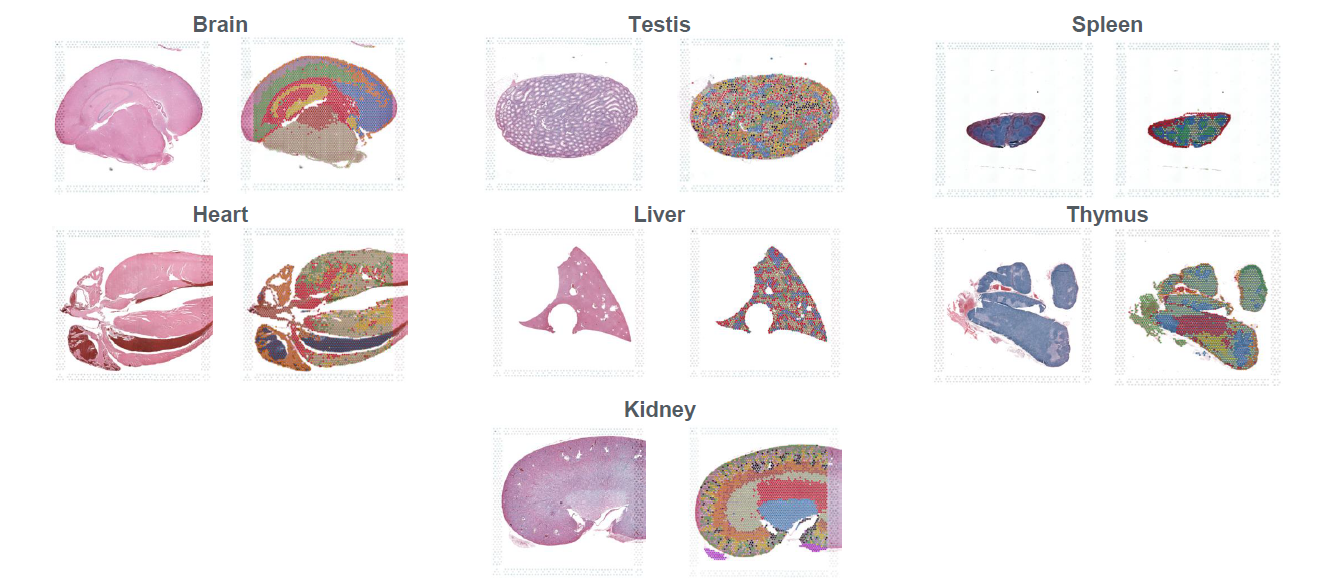
Figura 5. Ejemplos de Visium FFPE para tejido de ratón
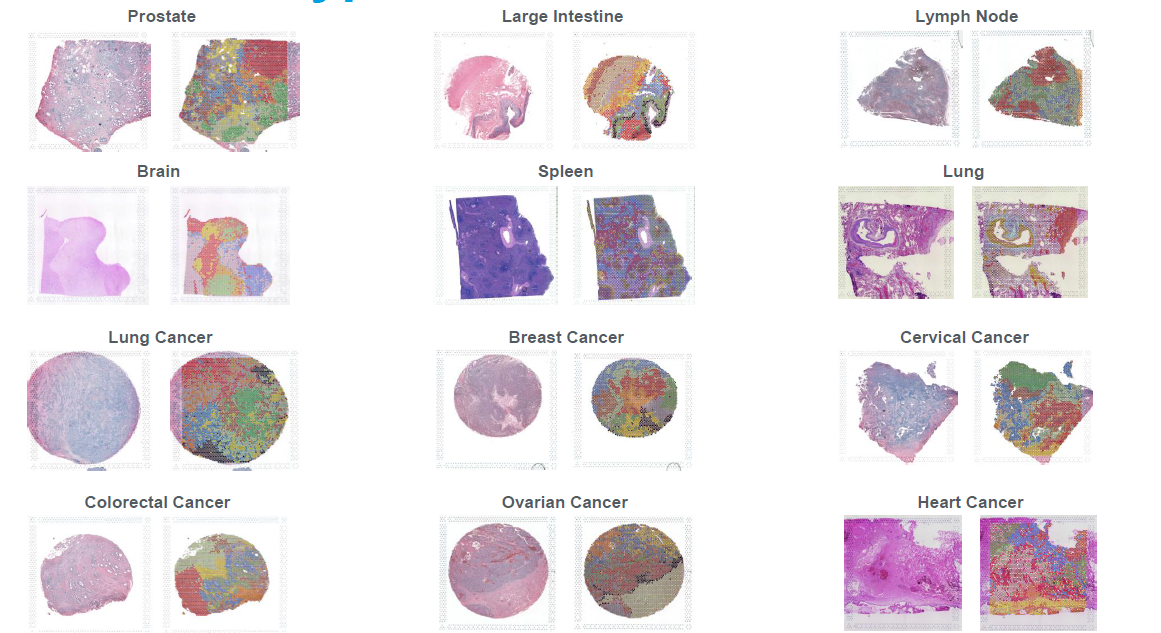
Figura 6. Ejemplos de Visium FFPE para tejido humano
La solución Visium para FFPE ya está disponible para Pre-Order, y desde Bonsailab os mantendremos informado de todos los detalles y novedades. Para más información: https://www.10xgenomics.com/products/spatial-gene-expression